6.2 聚类分析 EMP_cluster_analysis
聚类分析属于机器学习中非监督学习的范畴,其不需要预先设置分组,而是根据数据本身的特性研究分类方法,并遵循这个分类方法对数据进行合理的分类,最终将相似数据分为一组,从而帮助用户发现数据的模式和结构,识别离群值。
6.2.1 根据assay对样本进行聚类分析
🏷️示例:
MAE |>
EMP_assay_extract(experiment = 'geno_ec') |>
EMP_cluster_analysis()
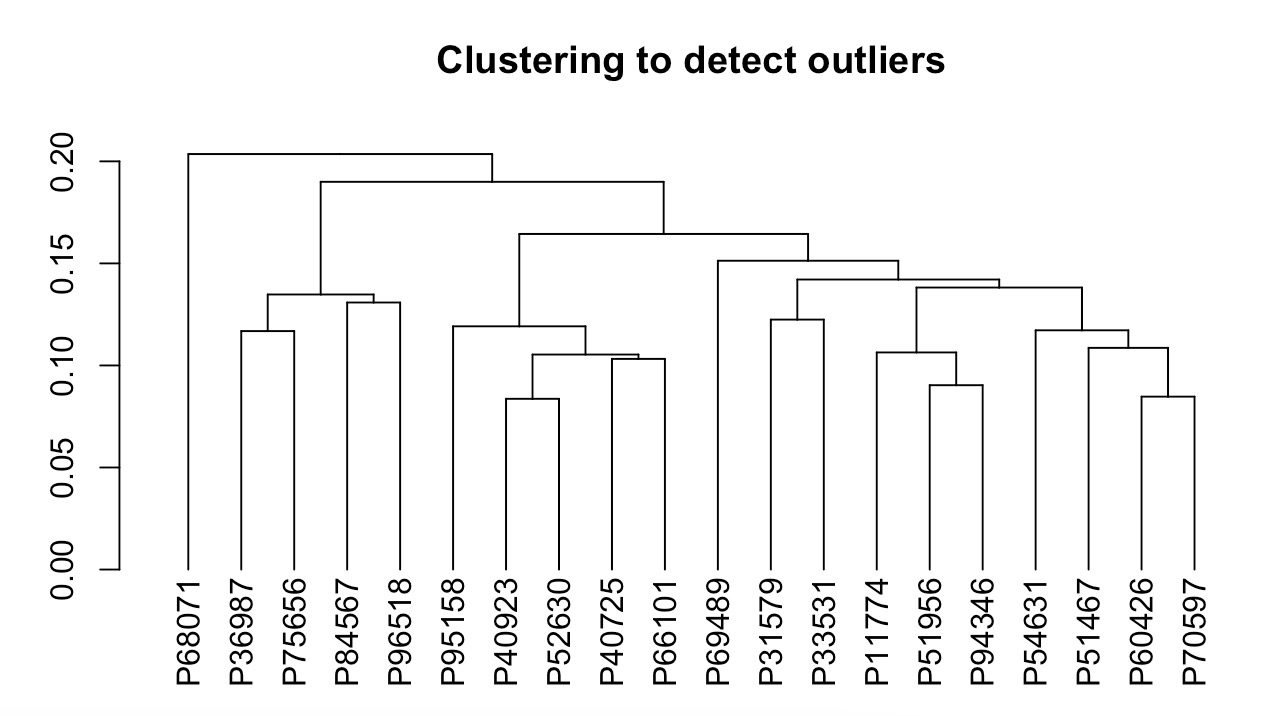
可以通过指定参数h,进行聚类分组标记。
MAE |>
EMP_assay_extract(experiment = 'geno_ec') |>
EMP_cluster_analysis(h=0.15)
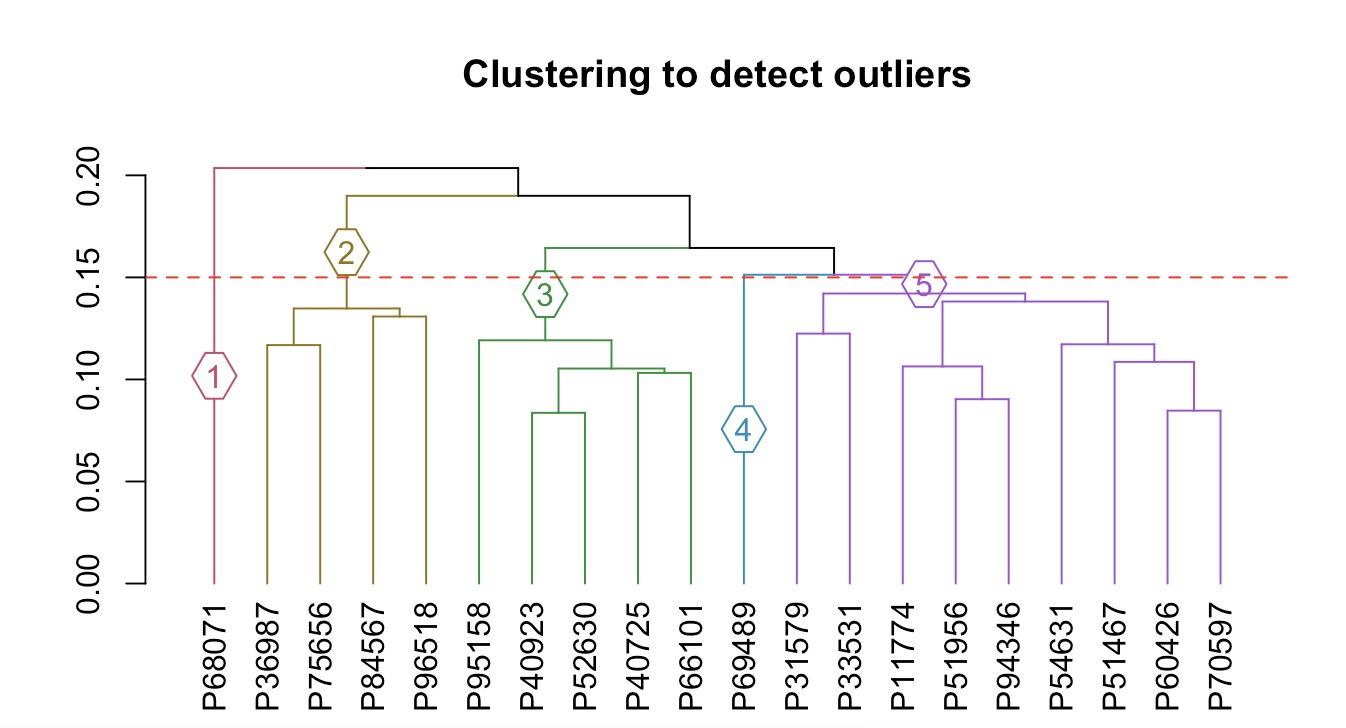
使用模块EMP_filter对分组为1和4的离群样本进行过滤。
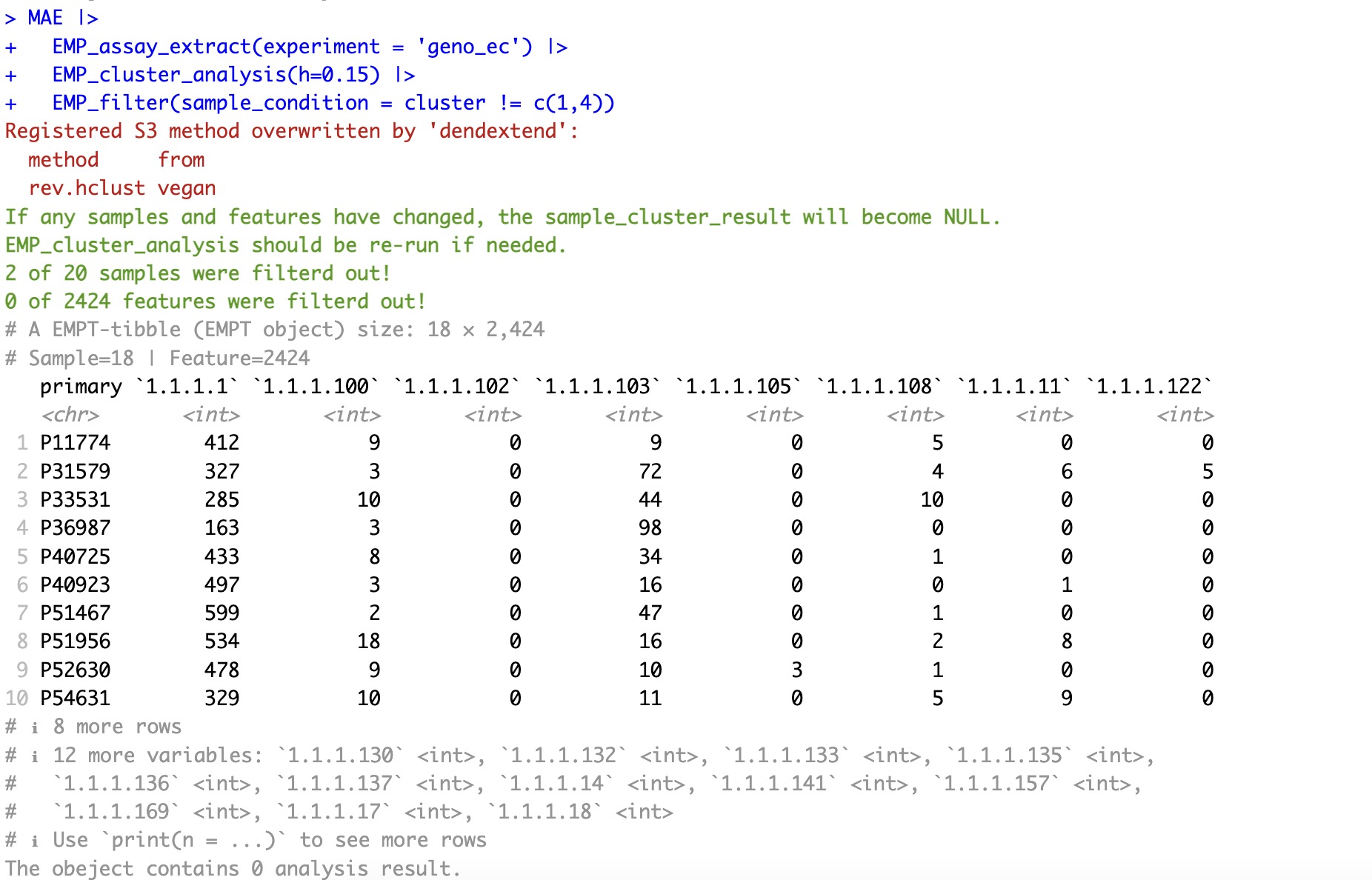
MAE |>
EMP_assay_extract(experiment = 'geno_ec') |>
EMP_cluster_analysis(h=0.15) |>
EMP_filter(sample_condition = cluster != c(1,4))
6.2.2 根据assay对特征进行聚类分析
EMP_cluster_analysis也可以对特征进行聚类分析,以快速发现感兴趣的特征之间的层级关系。
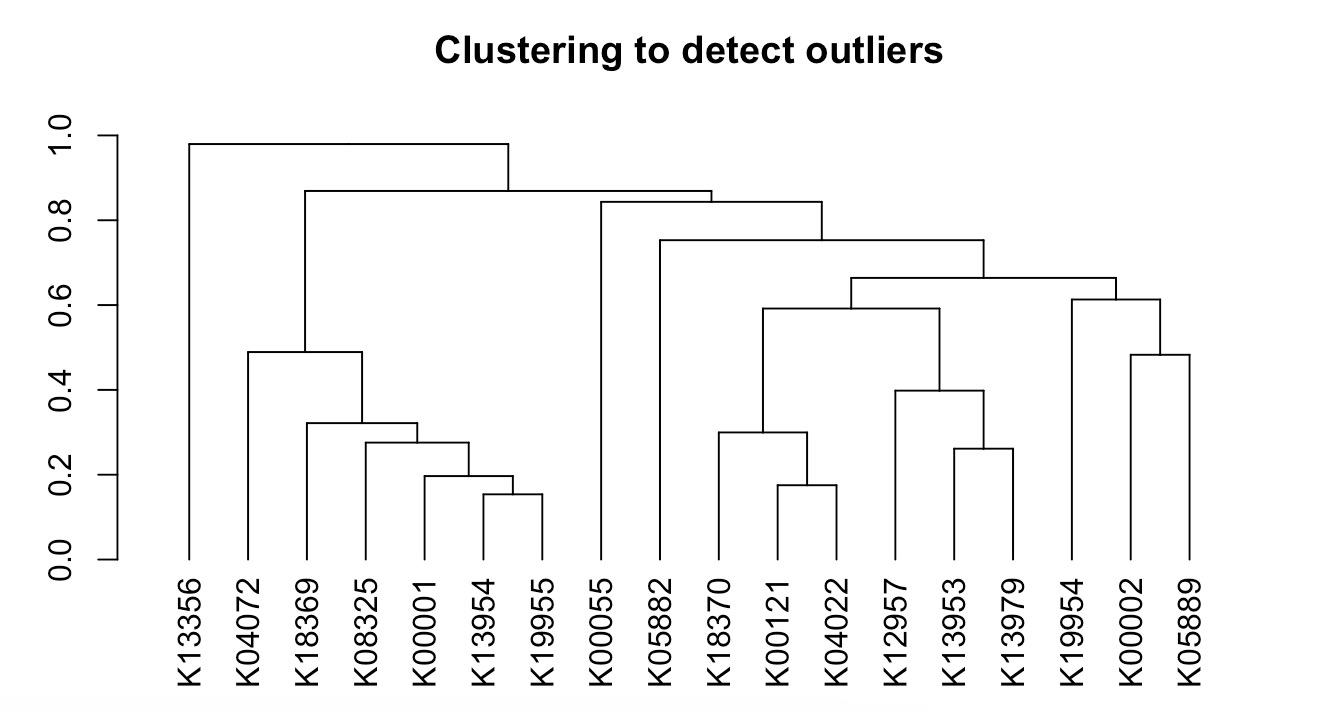
🏷️示例:查找酒精相关的KO基因,对特征进行聚类分析。
注意:
本示例是通过查找rowdata的Name列中的字符串,以确定包含alcohol的KO基因,而并非根据知识图谱确定的与跟酒精相关的基因。
本示例是通过查找rowdata的Name列中的字符串,以确定包含alcohol的KO基因,而并非根据知识图谱确定的与跟酒精相关的基因。
MAE |>
EMP_assay_extract(experiment='geno_ko',
pattern = 'alcohol',pattern_ref = 'Name') |>
EMP_cluster_analysis(rowdata = T)
6.2.3 根据coldata对样本进行聚类分析
在临床数据分析中,受试者的样本相关数据通常包含基本信息(例如:饮食数据、量表数据等)。模块EMP_colda_extract可以通过指定参数action='add'将coldata转换成assay,从而进行下游分析。
🏷️示例:
注意:
①当coldata样本相关数据较多时,可以使用参数
②当因部分特征的缺失值导致计算部分样本间距离为缺失时,将自动取最大默认值1。用户可以使用参数
①当coldata样本相关数据较多时,可以使用参数
coldata_to_assay选择所需要转换的样本相关数据。如果未指定,则默认将coldata中全部的连续型变量转换为assay。②当因部分特征的缺失值导致计算部分样本间距离为缺失时,将自动取最大默认值1。用户可以使用参数
pseudodist修改默认值,或者使用EMP_impute模块在聚类分析前插补缺失值。
MAE |>
EMP_coldata_extract(action = 'add',
coldata_to_assay = c('SAS','SDS','HAMA','HAMD','PHQ9','GAD7')) |>
EMP_cluster_analysis()
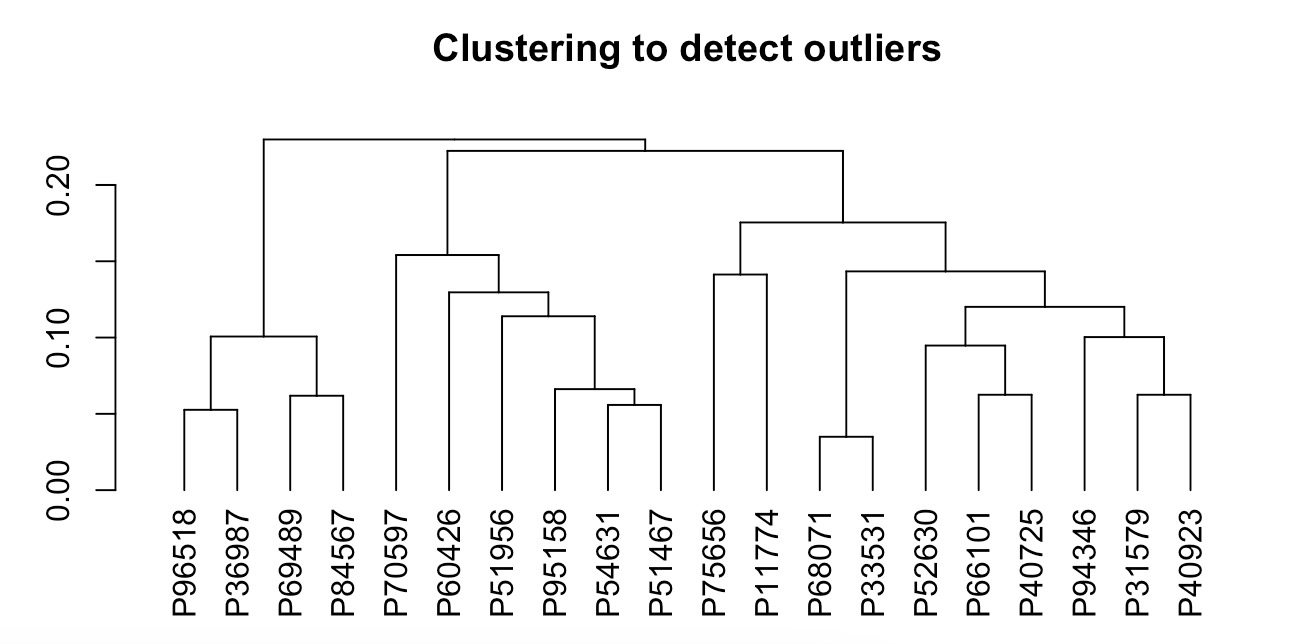
MAE |>
EMP_coldata_extract(action = 'add',
coldata_to_assay = c('SAS','SDS','HAMA','HAMD','PHQ9','GAD7')) |>
EMP_cluster_analysis(rowdata=TRUE)
